의료 영상 이미지의 특징
- 의료 영상 이미지는 개인 정보 보호 등의 이슈로 인해 데이터를 구하는 것이 어려움
- 라벨링 작업 자체가 전문적 지식을 요하므로 데이터셋 구축에 비용이 큼
- 희귀질병을 다루는 경우 데이터를 입수하는 것 자체가 어려움
- 음성/양성 데이터 간 imbalance가 심합니다. 학습에 주의가 필요합니다.
- 이미지만으로 진단이 쉽지 않아 다른 데이터와 결합해서 해석해야 할 가능성↑
의료 영상 종류
X-Ray
X-RAY는 방사선의 일종으로 지방, 근육, 천, 종이같이 밀도가 낮은 것은 수월하게 통과하지만, 밀도가 높은 뼈, 금속 같은 물질은 잘 통과할 수 없음
CT
- 환자를 중심으로 X-RAY를 빠르게 회전하여 3D 이미지를 만들어내는 영상
- 신체의 단면 이미지를 "Slice"라고 합니다. 이러한 Slice는 단층 촬영 이미지라고도 하며 기존의 X-RAY보다 더 자세한 정보를 포함
MRI
MRI는 Magnetic Resonance Imaging(자기 공명 영상)의 줄임말로 신체의 해부학적 과정과 생리적 과정을 보기 위해 사용하는 의료 영상 기술
MRI 스캐너는 강한 자기장를 사용하여 신체 기관의 이미지를 생성하고 MRI는 CT, X-RAY와 다르게 방사선을 사용하지 않아서 방사선의 위험성에서는 보다 안전
의료영상 자세 분류
- Sagittal plane : 시상면. 사람을 왼쪽과 오른쪽을 나누는 면.
- Coronal plane : 관상면. 인체를 앞뒤로 나누는 면.
- Transverse plane : 횡단면(수평면). 인체를 상하로 나누는 면.
데이터에서 주로 다룰 부분은 관상면
X-Ray 특성
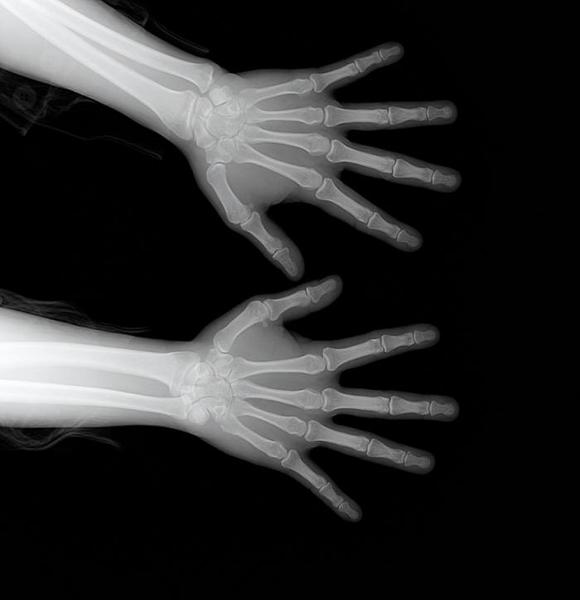
- 뼈 : 하얀색
- 근육 및 지방 : 연한 회색
- 공기 : 검은색

몸을 통과한 전자기파에 따라 다르게 나오는 명암
폐렴에 대한 이해
폐렴(pneumonia 뉴모니아)은 폐에 염증이 생긴 상태로 중증의 호흡기 감염병이다. 세균을 통한 감염이 가장 많으며, 바이러스, 균류, 또는 기타 미생물도 원인이 될 수가 있다. 드물게는 알레르기 반응이나 자극적인 화학 물질을 흡입해 발생하기도 한다. 노인이나 어린아이, 혹은 전체적으로 상태가 안 좋은 환자들이나 기침 반사가 약한 사람들에게는 흡인성 폐렴이 발생한다. 그리고 세균이 원인인 경우는 항생제로 치료를 할 수 있다. 항생제가 생기기 전에는 50~90%가 사망할 정도로 위험한 질환이었으나, 현재는 거의 사망하지 않는다. 1940년대에 항생제가 개발되기 전까지는 폐렴 환자의 1/3 정도가 사망하였다. 오늘날에는 적절한 의학적 치료로 폐렴 환자의 95% 이상이 회복된다. 그러나 일부 저개발국(개발 도상국)에서는 폐렴이 여전히 주요 사망 원인 중 하나이다.
출처 : https://ko.wikipedia.org/wiki/폐렴
염증은 유해한 자극에 대한 생체반응 중 하나로 면역세포, 혈관, 염증 매개체들이 관여하는 보호반응이다. 염증의 목적은 세포의 손상을 초기 단계에서 억제하고, 상처 부분의 파괴된 조직 및 괴사된 세포를 제거하며, 동시에 조직을 재생하는 것이다.
데이터 처리
데이터는 이 링크에서도 받아오실 수 있습니다
Chest X-Ray Images (Pneumonia)
5,863 images, 2 categories
www.kaggle.com
# 캐글 API를 통해 dataset을 불러옵니다
# 사용 환경이 코랩임을 참고하세요
!kaggle datasets download -d paultimothymooney/chest-xray-pneumonia
!unzip /content/chest-xray-pneumonia.zip# 라이브러리 import
import os, re
import random, math
import numpy as np
import tensorflow as tf
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings(action='ignore')
# 아래는 tpu 사용가능 시 넣어야 할 코드입니다만, 그냥 사용시
# 데이터셋 만드는 과정에서 에러 발생합니다
#tpu = tf.distribute.cluster_resolver.TPUClusterResolver()
#tf.config.experimental_connect_to_cluster(tpu)
#tf.tpu.experimental.initialize_tpu_system(tpu)
#strategy = tf.distribute.experimental.TPUStrategy(tpu)
#BATCH_SIZE = 16 * strategy.num_replicas_in_sync데이터의 중복 확인
print(len(os.listdir('/content/chest_xray/train/NORMAL')))
print(len(os.listdir('/content/chest_xray/chest_xray/train/NORMAL')))
print(len(os.listdir('/content/chest_xray/__MACOSX/chest_xray/train/NORMAL')))
#result
1341
1342
1342똑같인 chest_xray의 파일명이 여러개가 있었습니다 혹여 의심스러워서 하나를 테스트 해봤습니다
is_duplicated = {}
for i in os.listdir('/content/chest_xray/chest_xray/train/NORMAL'):
if i in os.listdir('/content/chest_xray/train/NORMAL'):
is_duplicated[i] = True
else:
is_duplicated[i] = False중복이 확인되어서 다른 chest_xray 파일은 사용하지 않기로 했습니다
다음으로 주요 변수들을 지정합니다
AUTOTUNE = tf.data.experimental.AUTOTUNE
IMAGE_SIZE = [720,720]
ROOT_PATH = '/content/chest_xray'
TRAIN_PATH = ROOT_PATH + '/train/*/*.jpeg' #jpeg 형태의 데이터를 모두 지정합니다
VAL_PATH = ROOT_PATH + '/val/*/*.jpeg' #폴더내에 jpg형태의 파일이 섞여있었습니다
TEST_PATH = ROOT_PATH + '/test/*/*.jpeg'tensorflow의 io메서드를 활용해서 폴더내의 데이터를 바로 load 할 수 있습니다
추후에 tf.Dataset의 형식을 만들기까지 영향을 미칩니다
train_files = tf.io.gfile.glob(TRAIN_PATH)
test_files = tf.io.gfile.glob(TEST_PATH)
val_files = tf.io.gfile.glob(VAL_PATH)
print(len(train_files),len(test_files),len(val_files))
#result
5216 624 16
# valid 파일의 갯수가 얼마 없어 train data와 합쳐서 다시 나눕니다
filenames = tf.io.gfile.glob(TRAIN_PATH)
filenames.extend(tf.io.gfile.glob(VAL_PATH))sanity check
filenames[:10]
#result
['/content/chest_xray/train/NORMAL/NORMAL2-IM-0882-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-0930-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-0521-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-1301-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-1045-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-1112-0001.jpeg',
'/content/chest_xray/train/NORMAL/IM-0362-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-1294-0001-0001.jpeg',
'/content/chest_xray/train/NORMAL/IM-0697-0001.jpeg',
'/content/chest_xray/train/NORMAL/NORMAL2-IM-0522-0001.jpeg']폴더 경로에 따라 양성과 음성 데이터가 구분되어 있기 때문에 파일명으로 각 데이터의 수를 파악합니다
COUNT_NORMAL = len([filename for filename in train_filenames if "NORMAL" in filename])
print(f"Normal images count in training set: {COUNT_NORMAL}")
COUNT_PNEUMONIA = len([filename for filename in train_filenames if "PNEUMONIA" in filename])
print(f"Pneumonia images count in training set: {COUNT_PNEUMONIA}")
#result
Normal images count in training set: 1111
Pneumonia images count in training set: 3074합쳤던 train과 val을 섞은 뒤 비율에 맞게 분리합니다(80%, 20%)
train_size = int((len(filenames)*0.8))
random.seed(8)
random.shuffle(filenames)
train_filenames = filenames[:train_size]
val_filenames = filenames[train_size:]
print(len(train_filenames),len(val_filenames))tensorflow의 from_tensor_slices를 사용하면 쉽게 tensorflow의 dataset 형태로 만들 수 있습니다
train_list_ds = tf.data.Dataset.from_tensor_slices(train_filenames)
val_list_ds = tf.data.Dataset.from_tensor_slices(val_filenames)
#file count
TRAIN_IMG_COUNT = tf.data.experimental.cardinality(train_list_ds).numpy()
print(TRAIN_IMG_COUNT)
VAL_IMG_COUNT = tf.data.experimental.cardinality(val_list_ds).numpy()
print(VAL_IMG_COUNT)
#result
4185
1047dataset의 전처리를 위한 함수를 만들고, map method를 통해 빠르게 전처리를 적용할 수 있습니다
이때 주석처리된 with open을 사용하면 에러가 발생합니다
TPU를 사용하고자 시도하던 과정인데 TPU는 tf.io 방식과 에러가 있어서 다른 읽기 방식을 시도했던 것인데, with open 형식은 데이터셋의 작동과는 맞지 않아 에러가 발생합니다
def get_label(file_path):
parts = tf.strings.split(file_path,os.path.sep) # path를 '/' 기준 split
return parts[-2] == "PNEUMONIA"
def decode_img(img):
img = tf.image.decode_jpeg(img, channels=3) #이미지 uint8 tensor로 수정
img = tf.image.convert_image_dtype(img, tf.float32)
img = tf.image.resize(img, IMAGE_SIZE) # 이미지 사이즈를 IMAGE_SIZE만큼 수정
return img
def process_path(file_path):
label = get_label(file_path)
img = tf.io.read_file(file_path) # 이미지 읽기
#with open(file_path, 'rb') as f:
# img = f.read() # get label
img = decode_img(img) # image preprocess
return img, label
train_ds = train_list_ds.map(process_path, num_parallel_calls=AUTOTUNE)
val_ds = val_list_ds.map(process_path, num_parallel_calls=AUTOTUNE)sanity check
for image, label in train_ds.take(1):
print("Image shape: ", image.numpy().shape)
print("Label: ", label.numpy())
Image shape: (720, 720, 3)
Label: Falsetest set도 동일한 과정의 전처리를 진행합니다
test_list_ds = tf.data.Dataset.list_files(TEST_PATH)
TEST_IMAGE_COUNT = tf.data.experimental.cardinality(test_list_ds).numpy()
test_ds = test_list_ds.map(process_path, num_parallel_calls=AUTOTUNE)
test_ds = test_ds.batch(BATCH_SIZE)
print(TEST_IMAGE_COUNT)
#result
624만들어진 데이터셋들에 BATCH를 생성합니다
def prepare_for_training(ds, shuffle_buffer_size=1000):
ds = ds.shuffle(buffer_size=shuffle_buffer_size)
ds = ds.repeat()
ds = ds.batch(BATCH_SIZE)
ds = ds.prefetch(buffer_size=AUTOTUNE)
return ds
train_ds = prepare_for_training(train_ds)
val_ds = prepare_for_training(val_ds)sanity check
# 이미지 배치를 입력하면 여러장의 이미지를 보여줍니다.
def show_batch(image_batch, label_batch):
plt.figure(figsize=(10,10))
for n in range(BATCH_SIZE):
ax = plt.subplot(4,math.ceil(BATCH_SIZE/4),n+1)
plt.imshow(image_batch[n])
if label_batch[n]:
plt.title("PNEUMONIA")
else:
plt.title("NORMAL")
plt.axis("off")
image_batch, label_batch = next(iter(train_ds))
show_batch(image_batch.numpy(), label_batch.numpy())데이터의 불균형 확인
딥러닝에서 클래스 불균형을 다루는 방법
딥러닝에서 클래스 불균형을 다루는 방법 현실 데이터에는 클래스 불균형 (class imbalance) 문제가 자주 있다. 어떤 데이터에서 각 클래스 (주로 범주형 반응 변수) 가 갖고 있는 데이터의 양에 차이
3months.tistory.com
weight_for_0 = (1 / COUNT_NORMAL)*(TRAIN_IMG_COUNT)/2.0
weight_for_1 = (1 / COUNT_PNEUMONIA)*(TRAIN_IMG_COUNT)/2.0
class_weight = {0: weight_for_0, 1: weight_for_1}
print('Weight for NORMAL: {:.2f}'.format(weight_for_0))
print('Weight for PNEUMONIA: {:.2f}'.format(weight_for_1))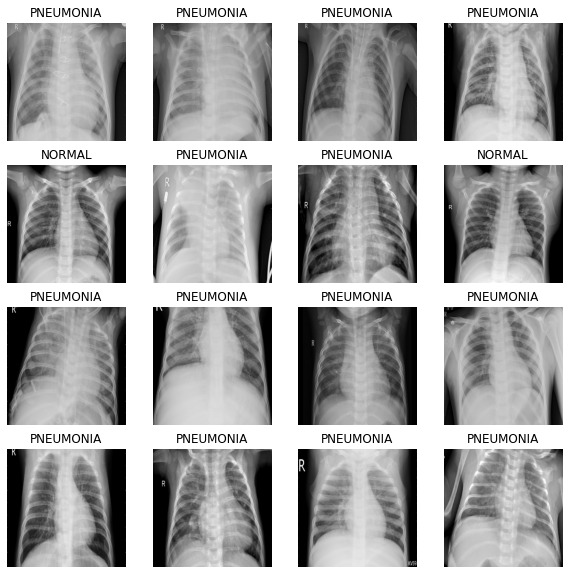
모델 구성
모델 구성에서 convblock과 denseblock에서 batch_normalization과 dropout이 동시에 적용되는 부분이
나오는데요, 통상적으로는 사용 안될 방법으로서 나오지만 링크에서 자세한 내용을 볼 수 있습니다
https://arxiv.org/pdf/1905.05928.pdf
모델을 구성합니다
def conv_block(filters):
block = tf.keras.Sequential([
tf.keras.layers.SeparableConv2D(filters, 3, activation='relu',padding='same'),
tf.keras.layers.SeparableConv2D(filters, 3, activation='relu',padding='same'),
tf.keras.layers.BatchNormalization(),
tf.keras.layers.MaxPool2D()
])
return block
def dense_block(units, dropout_rate):
block = tf.keras.Sequential([
tf.keras.layers.Dense(units, activation='relu'),
tf.keras.layers.BatchNormalization(),
tf.keras.layers.Dropout(dropout_rate)
])
return block
def build_model():
model = tf.keras.Sequential([
tf.keras.Input(shape=(IMAGE_SIZE[0], IMAGE_SIZE[1], 3)),
tf.keras.layers.Conv2D(16, 3, activation='relu',padding='same'),
tf.keras.layers.Conv2D(16, 3, activation='relu',padding='same'),
tf.keras.layers.MaxPool2D(),
conv_block(32),
conv_block(64),
conv_block(128),
tf.keras.layers.Dropout(0.2),
conv_block(256),
tf.keras.layers.Dropout(0.2),
tf.keras.layers.Flatten(),
dense_block(512,0.7),
dense_block(128, 0.5),
dense_block(64, 0.3),
tf.keras.layers.Dense(1, activation='sigmoid')
])
return model
with tf.device('/GPU:0'):
model = tf.keras.Sequential([
tf.keras.Input(shape=(IMAGE_SIZE[0], IMAGE_SIZE[1], 3)),
tf.keras.layers.Conv2D(16, 3, activation='relu',padding='same'),
tf.keras.layers.Conv2D(16, 3, activation='relu',padding='same'),
tf.keras.layers.MaxPool2D(),
conv_block(32),
conv_block(64),
conv_block(128),
tf.keras.layers.Dropout(0.2),
conv_block(256),
tf.keras.layers.Dropout(0.2),
tf.keras.layers.Flatten(),
dense_block(512,0.7),
dense_block(128, 0.5),
dense_block(64, 0.3),
tf.keras.layers.Dense(1, activation='sigmoid')
])
METRICS = [
'accuracy',
tf.keras.metrics.Precision(name='precision'),
tf.keras.metrics.Recall(name='recall')
]
model.compile(
optimizer='adam',
loss='binary_crossentropy',
metrics=METRICS
)
#call back function
es = tf.keras.callbacks.EarlyStopping(
monitor='val_loss',patience=2
)
with tf.device('/GPU:0'):
history = model.fit(
train_ds,
steps_per_epoch=TRAIN_IMG_COUNT // BATCH_SIZE,
epochs=5,
callbacks=[es],
validation_data=val_ds,
validation_steps=VAL_IMG_COUNT // BATCH_SIZE,
class_weight=class_weight,
)배치가 클 경우에는 학습과 로스가 크게 왔다갔다 하기 때문에 early stop을 걸더라도 이미 loss가 많이 올라간 상태에서 종료될 수 있습니다 기존의 128~256배치에서 16의 배치로 줄인 후에 가장 좋은 accuracy를 가질 수 있었습니다
위의 코드에서 사진의 해상도를 올리거나, data augment를 통해 좌우 위아래 반전을 추가하거나, Dense 증가 및 삭제, convblock 추가 및 삭제 등을 진행해봤지만 크게 효과를 볼 순 없었습니다
learning rate나 기타 하이퍼 파라미터도 건드릴 수 없는 마의 90프로 정확도의 영역 이후에 추가로 도전해볼 예정입니다
'23년 이전 글 > 모두의연구소 아이펠' 카테고리의 다른 글
| -27일차- 딥러닝 레이어의 이해 (0) | 2022.02.07 |
|---|---|
| -26일차- Deep Network (0) | 2022.02.04 |
| -23일차- Text Summarization (0) | 2022.01.27 |
| -22일차- 비지도 학습 (0) | 2022.01.26 |
| -21일차- 조금은 큰 고양이와, 맹수와 인셉션 댕댕이 (0) | 2022.01.25 |
